使用机器学习预测治疗反应
在临床试验中进行高通量分子测量变得越来越普遍。结合机器学习的进步,这些发展为基于大数据训练的算法开辟了预测患者是否会从治疗中获益的可能性。在这里,我们介绍培训这些算法的关键概念和常见方法。
关键点
- 将机器学习应用于临床试验数据可以实现生物标志物发现和算法定制治疗。
- 预测模型的复杂度各不相同:简单的方法(如逻辑回归)易于解释,并适用于小样本大小,而复杂的方法(如深度学习)更为强大,但需要大样本量。中间方法(如随机森林)在两者之间取得平衡。
- 过度拟合或模型对未经过训练的数据的泛化能力差是机器学习中的一个核心挑战。特征选择,模型正则化和交叉验证用于避免过度拟合。
- 曲线下面积(AUC)是预测模型性能的常见且可靠的度量,除了ROC曲线之外,ROC曲线突出了灵敏度和特异性之间的平衡。
- 预测模型的复杂性以及在临床设置中量化其生物标志物的可行性影响着模型的临床适用性。
介绍
将机器学习应用于临床数据的目的是建立一个计算模型,以患者特异方式从输入变量(任何可用的人口统计,临床和分子数据)中预测目标变量(如治疗反应)。除了预测模型,这样的分析还回答了以下关键问题:
-
输入变量能够多好地预测治疗反应?
-
哪些输入变量是有预测能力的生物标志物?
我们的重点是预测治疗反应,但这同样适用于预测其他临床变量,例如患者生存。
训练模型
通常,目标变量被视为二元变量,例如,0表示未响应,1表示响应治疗。模型是以算法方式训练的,根据输入变量预测目标变量。
在训练阶段,算法使用输入变量和目标变量来建模它们之间的关系(见下图)。在训练阶段使用的任何数据都称为训练数据。模型训练后,可以仅使用输入变量来预测目标变量。重要的是,可以在验证数据上估计模型的性能,而这些数据未用于训练。
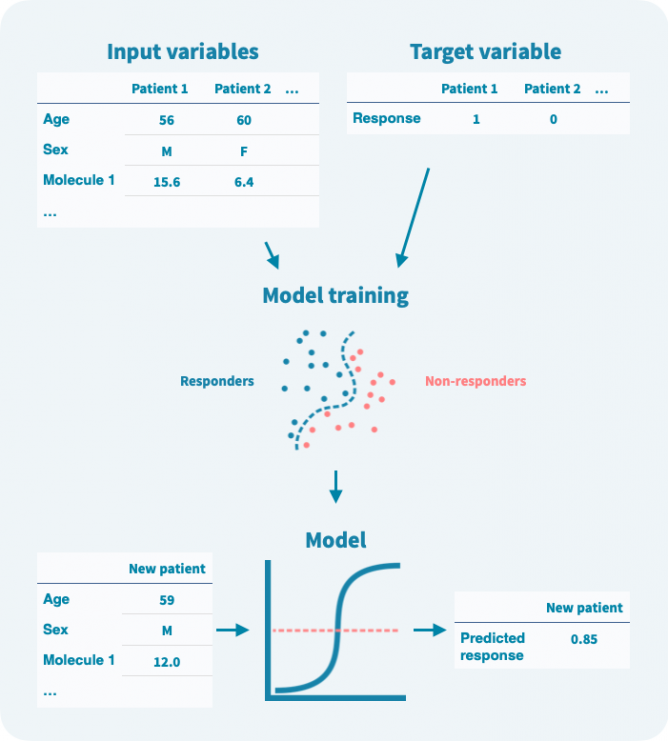
即使目标变量是二元的,模型预测通常也是概率性的——在0和1之间的数字。可以定义一个阈值来将预测二元化。设置阈值涉及灵敏度和特异性之间的权衡(参见模型验证和性能评估部分)。
输入变量可以进行预过滤以减少噪声、模型复杂度和计算量。这种预过滤称为特征选择(被认为是无信息量的变量被过滤掉,而信息量丰富的变量被选定)。此外,在训练阶段可以通过正则化来限制模型的复杂度,或者逐步惩罚算法增加模型的复杂度。限制模型的复杂度旨在避免过度拟合,即模型过度学习了训练数据:它在训练数据上表现准确,但在验证数据上表现不佳。
模型类型
用于从输入变量预测目标变量的计算模型具有多种形式,但模型选择中涉及的主要权衡与模型的复杂性有关。更复杂的模型可以实现更好的预测,但需要更多的数据,更难解释,并且更容易过拟合。我们可以根据其复杂性将模型类型分类如下表所示:

模型验证和性能评估
在量化预测模型的性能时,需要确保它不仅在训练数据上工作足够准确,而且也可能在未用于训练模型的新样本上工作。
假设我们已经训练了一个模型来预测治疗反应(介于0和1之间的值),并且我们使用已知反应的十个患者的输入变量运行该模型。通过使用任何阈值对预测进行二元化,例如0.5,我们可以将十个患者的预测和已知反应制成混淆矩阵,看起来像这样:

预测算法或分类器最常见的性能指标基于混淆矩阵。在上面的例子中,10名患者中有7名治疗反应被正确预测,总准确率为70%。假阳性率(FPR)为4中的1,即25%,假阴性率(FNR)为6中的2,即33%。此外,可以计算真阳性率(TPR或灵敏度)为1−FNR = 67%,真阴性率(TNR或特异度)为1−FPR = 75%。
尽管总体准确率是模型性能的简单易懂的度量方式,但问题在于它需要定义一个硬阈值来将预测二值化。一个更健壮的性能指标,它不依赖于单个阈值值,称为曲线下面积(AUC),并且是基于ROC曲线计算的。ROC曲线显示真阳性率如何取决于假阳性率(见下图)。
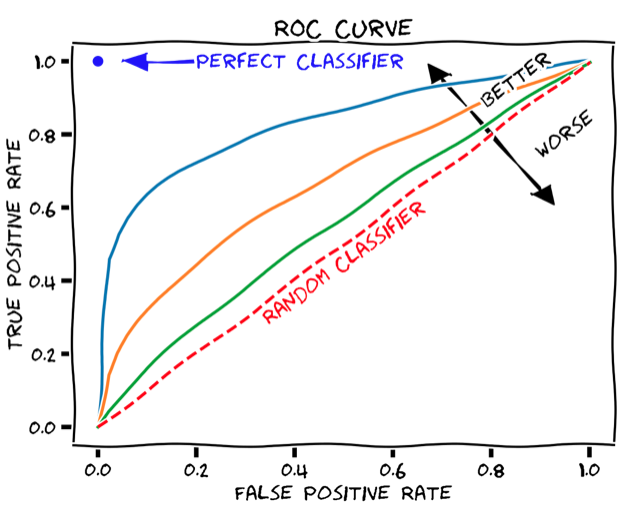
ROC曲线可以通过逐渐增加二元分类阈值从0(其中所有患者都被分类为响应者,TPR和FPR均为1,即ROC图的右上角)到1(其中所有患者都被分类为非响应者,TPR和FPR均为0,即ROC图的左下角)来产生。 AUC只是保留在ROC曲线下方的图形面积。任何分类器的AUC至少为0.5,对应于随机分类器的直线ROC曲线(上图中的虚线红线)。理想的、完美的分类器将具有最大的AUC值1。因此,现实世界的分类器的AUC值通常介于0.5和1之间。
理想情况下,应使用训练数据来训练模型,并有单独的验证队列来估计性能(最重要的是AUC),以确保模型具有可推广性。但是,对于样本数量有限的情况,通常采用交叉验证方法。在交叉验证中,模型使用(随机选择的)部分数据进行训练,并使用剩余数据进行验证,该过程重复进行,通常是使每个患者在训练中使用9次,验证模型1次(10倍交叉验证)。然后可以使用适当定义的分别训练的模型的平均值作为最终模型。
一种模型还是多种模型?
在任何实际的机器学习项目中,主要限制往往是数据量的大小。更大的样本量可以提供更好的模型,从中发现更好的生物标志物和更深入的生物学结论。将来自不同但相似的患者队列的数据(如果有)合并以训练单个模型是增加样本量的一种方式。如果存在多个系统性差异的变量(例如年龄、性别、使用的测量平台),这些变量可能会混淆:即使它们之间没有真正的关系,算法也可能将一个变量视为另一个变量的代理。在多个队列中,可以同时使用所有队列和每个队列单独进行训练模型。仔细检查模型之间的预测变量可能会揭示可能存在的混淆因素。
关于临床适用性的说明
除了模型的预测能力外,还有其他因素决定其有用性。其中之一是其复杂性:与复杂模型相比,简单模型更快,更容易作为易于使用的工具用于研究和临床目的。另一个因素是在临床环境中量化所需生物标志物的可行性。因此,将模型训练在体液样本数据上可能比在实体组织活检中发现需要侵入性样本收集的生物标志物更有意义。